Statistisches Praktikum im SS 2022
Prof. Dr. Gaby Schneider, unter Mitarbeit von Solveig Plomer und Fabian Roth
Seminar im SS 2022. Do. 12:30 Uhr, 711 groß.
Thema 1: Statistische Methoden zur Diabetes-Früherkennung
|
In Kooperation mit Prof. Dr. Viktor Krozer und Dr. Giacomo Ulisse,
Physikalisches Institut, Arbeitsgruppe Terahertz-Photonik,
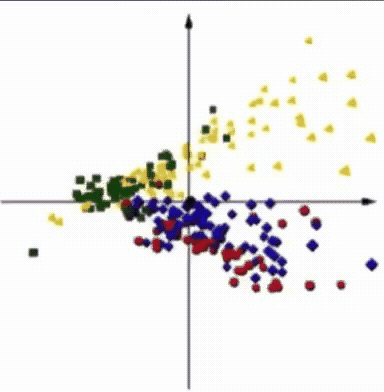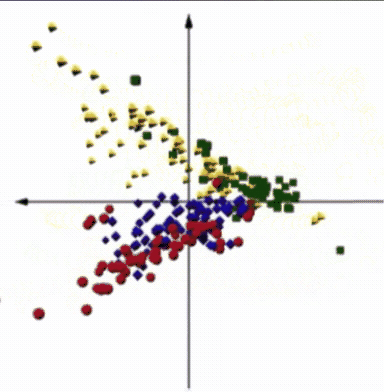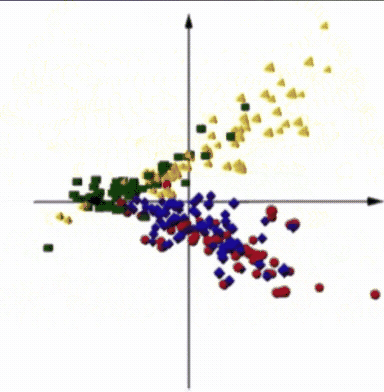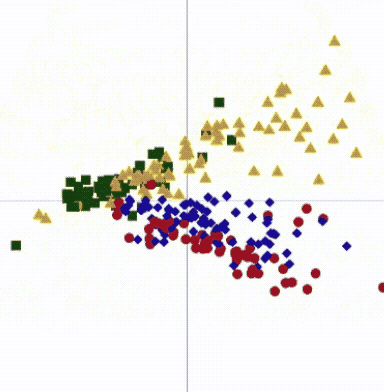
Goethe-Universität Frankfurt Nichtinvasive Diabetesmessungen mit Millimeterwellen-SpektrometerDie Daten für die nichtinvasive Untersuchungen an Mäusen wurden mit Hilfe eines Millimeterwellen-Spektrometers erhalten. Das Instrument misst den Durchgang und die Reflexion von elektromagnetischen Wellen durch eine Hautfalte. Das Spektrometer arbeitet derzeit im Frequenzbereich 75 - 110 GHz. |
  |